准备工作
#安装必要的R包
options("repos" = c(CRAN="http://mirrors.tuna.tsinghua.edu.cn/CRAN/"))
if(!require(tidyr))install.packages("tidyr",update = F,ask = F)
if(!require(dplyr))install.packages("dplyr",update = F,ask = F)
if(!require(stringr))install.packages('stringr',update = F,ask = F)
if(!require(tibble))install.packages('tibble',update = F,ask = F)
library(tidyr)
library(dplyr)
library(stringr)
library(tibble)
更换电脑时重新运行代码,发现R版本过期了,tidyr包需要R4.1.3以上,更新后无报错。
专题1.数据框操作
order() 函数用于排序
准备一下样本数据
test <- iris[c(1:2,51:52,101:102),] #根据行号抽取需要的数据 rownames(test) =NULL #将行号变量清空 test
Sepal.Length Sepal.Width Petal.Length Petal.Width Species 1 5.1 3.5 1.4 0.2 setosa 2 4.9 3.0 1.4 0.2 setosa 3 7.0 3.2 4.7 1.4 versicolor 4 6.4 3.2 4.5 1.5 versicolor 5 6.3 3.3 6.0 2.5 virginica 6 5.8 2.7 5.1 1.9 virginica
order(test$Sepal.Length) #根据第一列排序 test[order(test$Sepal.Length),] #根据排序结果取出数据,升序 test[order(test$Sepal.Length,decreasing = T),] 降序
Sepal.Length Sepal.Width Petal.Length Petal.Width Species 2 4.9 3.0 1.4 0.2 setosa 1 5.1 3.5 1.4 0.2 setosa 6 5.8 2.7 5.1 1.9 virginica 5 6.3 3.3 6.0 2.5 virginica 4 6.4 3.2 4.5 1.5 versicolor 3 7.0 3.2 4.7 1.4 versicolor
arrange() 函数排序
普通排序
arrange(test,Sepal.Length) #升序 arrange(test,desc(Sepal.Length)) #降序
按照A列排序,如果A列相同,按照B列排序
arrange(test,Sepal.Length, Sepal.Width) #length为A列,Width为B列
mutate() 函数新增列
需重新定赋值原本变量,否则无改变
#两种方式赋值新的一列 test <- mutate(test,new= test$Sepal.Length* test$Sepal.Width) test$new <- test$Sepal.Length* test$Sepal.Width
select() 和 filter() 筛选函数
优秀的管道符号
x1 = filter (iris,Sepal.Width >3)
x2 = select (x1,c("Sepal.Length","Sepal.Width"))
x3 = arrange(x2,Sepal.Length)
# 加入管道符号连接,理解为向后传递
x = iris %>%
filter (Sepal.Width >3) %>%
select (c("Sepal.Length","Sepal.Width")) %>%
arrange(Sepal.Length)
专题2.表达矩阵画箱线图
生成一个固定不变的矩阵,做一些微调
set.seed(10086)
exp = matrix(rnorm(18),ncol = 6)
exp = round(exp,2) # 取几位小数
rownames(exp) = paste0("gene",1:3) #添加行名
colnames(exp) = paste0("test",1:6) #添加列名
exp[,1:3] = exp[,1:3]+1 #给前三列+1
exp
test1 test2 test3 test4 test5 test6
gene1 1.55 1.49 1.80 -0.37 -1.82 -1.62
gene2 -1.74 0.37 2.08 2.11 -0.22 1.42
gene3 1.57 1.25 1.32 2.49 0.58 -0.81
library(tidyr)
library(tibble)
library(dplyr)
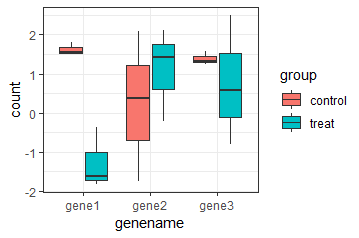
dat = t(exp) %>%
as.data.frame() %>%
rownames_to_column() %>%
mutate(group = rep(c("control","treat"),each = 3))
#增长
pdat = dat %>%
pivot_longer(cols = starts_with("gene")
names_to = "gene",
values_to = "count")
library(ggplot2) p = ggplot(pdat,aes(gene,count))+ geom_boxplot(aes(fill = group))+ theme_bw() p + facet_wrap(~gene,scales = "free")
专题3.连接
library(dplyr) inner_join(test1,test2,by="name") right_join(test1,test2,by="name") full_join(test1,test2,by="name") #全连接,取全集 semi_join(test1,test2,by="name") anti_join(test1,test2,by="name")